Pima Indian Diabetes 데이터 Clustering & Classification (Part.2)
- -
지난 글에 이어 모델 학습 및 평가를 해보겠습니다.
# 모델링
데이터에서 X와 y를 분리 시킵니다
#- Scaling -
스케일링은 예를들어 집값의 범위는 100억~천만원 단위인데 BMI 값은 18~30이라 모델링할때 수치가 큰쪽으로 치우쳐
모델링이 잘 안되는것을 방지하기위해 설정한 범위 내의 값으로 변환 시켜줍니다. scaling model로는 StandardScaler를 씁니다.
fit_transform()은 fit() 과 transform() 함께 수행하는 메소드 입니다.
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split
X = df.loc[:, df.columns != "Outcome"]
y = df.loc[:, df.columns == "Outcome"]
scaler = StandardScaler()
xScaled = scaler.fit_transform(X)
print(xScaled[:, :6])
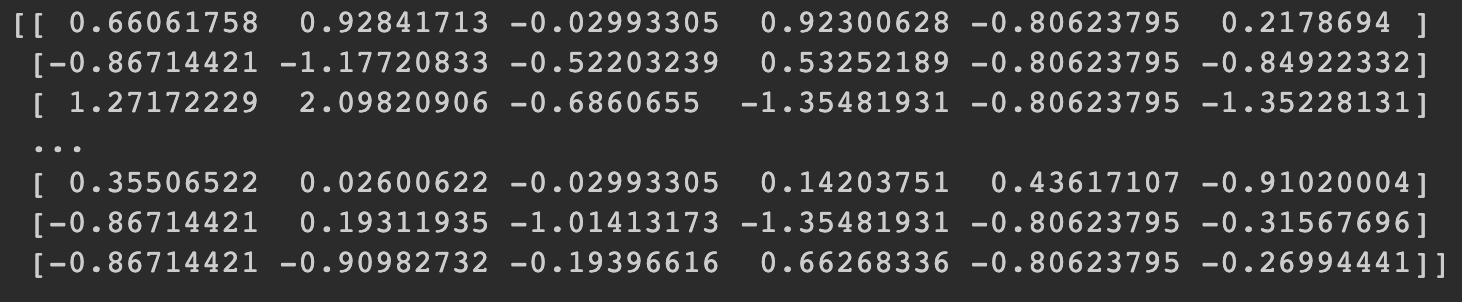
이제 모델 학습에 들어 갑니다.
사용할 모델입니다.
- LogisticRegression
- RandomForest
- DecisionTree
- SVM
#- GridSearchCV -
GridSearchCV는 최적의 파라미터를 찾아주고 교차검증도 해줍니다.
여기서 파라미터란 모델에서 bias 값 즉 예측할때 가장 적합한 값을 찾아준다고 보면됩니다.
y = wX+b 에서 b값이라고 보면됩니다.
#- SMOTE -
y의 값이 불균형적이라 1의 값을 늘리고 0의 값을 줄이고 하는 복합적으로 불균형을 균형있게
맞출 수 있도록 SMOTE를 씁니다. SMOTE는 데이터를 늘리고 줄여서 데이터를 변화시키기 때문에
반드시 train 데이터셋에만 적용합니다. test값은 실제로 테스트해봐야하기 때문에 그데로 보존
하여야 합니다.
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import GridSearchCV
def fitClasifiers(gs_clfs, X, y):
for clf in gs_clfs:
print(X.shape)
clf.fit(X, y)
def showGridsearchResult(gs_clfs):
estimators = []
scores = []
params = []
for clf in gs_clfs:
estimators.append(str(clf.estimator))
scores.append(clf.best_score_)
params.append(clf.best_params_)
for i, val in enumerate(estimators):
print(val)
print(scores[i])
print(params[i])
xTrain, xTest, yTrain, yTest = train_test_split(xScaled, y, test_size=0.2, stratify=y)
lr = LogisticRegression()
svc = SVC(probability=True)
dt = DecisionTreeClassifier()
rf = RandomForestClassifier()
# 혹시나 아래의 에러가 뜬다면
# ValueError: Solver lbfgs supports only 'l2' or 'none' penalties, got l1 penalty
# 아래의 코드처럼 penalty에 l2만 써주면 해결됩니다.
paramLr = {"penalty": ["l2"]}
#"linear", "poly", "rbf", "sigmoid" 에서 최적의 것을 찾아라는 의미입니다.
paramSvc = {"kernel": ["linear", "poly", "rbf", "sigmoid"]}
# max_depth는 파라미터를 3~6 사이로 최적의 값을 찾아라는 의미입니다.
paramTree = {"max_depth": [3, 4, 5, 6], "min_samples_split": [2, 3]}
# cv는 cross validation의 약자로 만약에 5로 설정하면 데이터가 100개 있다고 가정할때 20, 20, 20, 20, 20
# 씩 나누어 1~4번까지는 train data로 쓰고 나머지 5번은 test data로 씁니다.
# 이렇게 각 test data를 5번 돌리게 되므로 작은 데이터를 학습할때 좀 더 정확한 예측을 할 수 있습니다.
gsLr = GridSearchCV(lr, param_grid=paramLr, cv=5, refit=True)
gsSvc = GridSearchCV(svc, param_grid=paramSvc, cv=5, refit=True)
gsDt = GridSearchCV(dt, param_grid=paramTree, cv=5, refit=True)
gsRf = GridSearchCV(rf, param_grid=paramTree, cv=5, refit=True)
gsClfs = [gsLr, gsSvc, gsDt, gsRf]
# 혹시나 아래의 에러가 뜬다면
# DataConversionWarning: A column-vector y was passed when a 1d array was expected.
# Please change the shape of y to (n_samples,), for example using ravel().
# 아래의 코드처럼 y_train.values.ravel()으로 바꾸어 주면 해결됩니다.
fitClasifiers(gsClfs, xTrain, yTrain.values.ravel())
showGridsearchResult(gsClfs)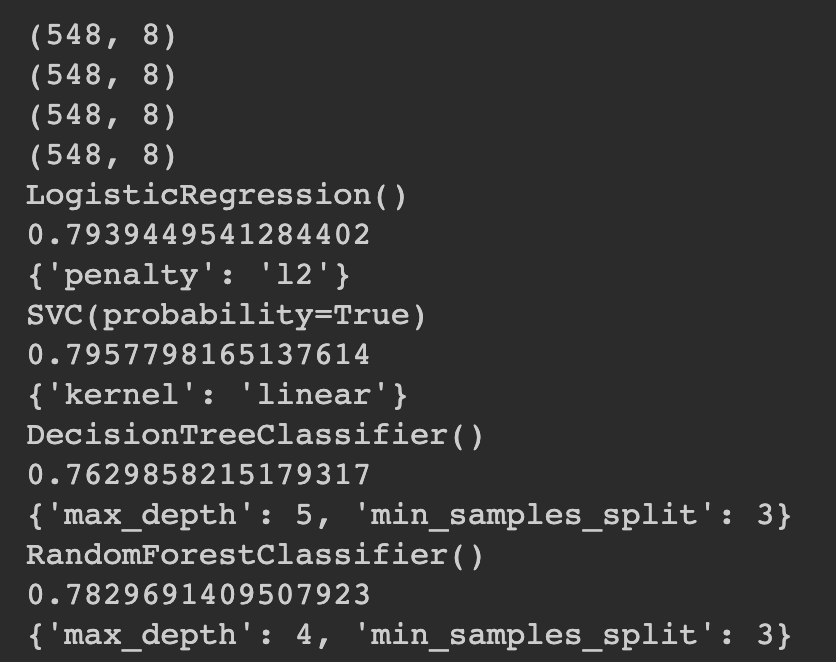
위의 결과는 acc 확률 입니다.
LogisticRegression acc : 79.3%
SVC acc : 79.5%
DecisionTreeClassifier acc : 76.2%
RandomForestClassifier : 78.2%
로 SVC가 가장 높게 나왔습니다. 하지만 acc로 모델 성능을 판단하기에는 위험이 있습니다.
예를들어 1000개의 데이터에서 900개의 y컬럼 데이터가 1이고 100개가 0일 경우, 모델이 그냥 1000번을 1이라고 예측을 하면
예측률은 90%가 되는 것입니다.
이런 평가방법을 방지하기 위해 우리는 roc curve와 roc_auc score.. 등 다른 평가지표를 알아야 합니다.
5. 평가지표들 살펴보기
- l2 패널티를 준 로지스틱 회귀 모델에서
- 혼동 행렬, 정확도, 재현률, 정밀도, roc curve와 roc_auc score 등을 살펴보자
머신러닝 성능 평가 - ROC 커브 개념 정리 링크
https://losskatsu.github.io/machine-learning/stat-roc-curve/#%EB%A8%B8%EC%8B%A0%EB%9F%AC%EB%8B%9D-%EC%84%B1%EB%8A%A5-%ED%8F%89%EA%B0%80---roc-%EC%BB%A4%EB%B8%8C-%EA%B0%9C%EB%85%90-%EC%A0%95%EB%A6%AC
from sklearn.metrics import accuracy_score, confusion_matrix, roc_auc_score
from sklearn.metrics import recall_score, precision_score, roc_curve
from sklearn.metrics import precision_recall_curve
def showMetrics(yTest, yPred):
confusion = confusion_matrix(yTest, yPred)
accuracy = accuracy_score(yTest, yPred)
precision = precision_score(yTest, yPred)
recall = recall_score(yTest, yPred)
print(confusion)
print("Acc : {}".format(accuracy))
print("precision : {}".format(precision))
print("recall : {}".format(recall))
# 아래와 같은 에러가 발생 한다면 패키지 버전 오류 때문에 그렇습니다.
# AttributeError: module 'seaborn' has no attribute 'lineplot'
# seaborn을 최신버전으로 업데이트 해줍니다.
def showPrecisionRecallCurve(yTest, probPositivePred):
precisions, recalls, thresholds = precision_recall_curve(yTest, probPositivePred)
print("th val : {}".format(thresholds[:4]))
print("precision val : {}".format(precisions[:4]))
print("recalls val : {}".format(recalls[:4]))
df = {
"thresholds": thresholds,
"precisions": precisions[:-1],
"recalls": recalls[:-1]
}
df = pd.DataFrame.from_dict(df)
sns.lineplot(x="thresholds", y="precisions", data=df)
sns.lineplot(x="thresholds", y="recalls", data=df)
plt.show()
def showRocCurve(yTest, probPositivePred):
fpr, tpr, thresholds = roc_curve(yTest, probPositivePred)
print("fpr val : {}".format(fpr[:4]))
print("tpr val : {}".format(tpr[:4]))
print("thresholds val : {}".format(thresholds[:4]))
df = {"threshold": thresholds, "fpr": fpr, "tpr": tpr}
df = pd.DataFrame.from_dict(df)
sns.lineplot(x="fpr", y="tpr", data=df)
plt.show()
rocScore = roc_auc_score(yTest, probPositivePred)
print("roc score : " + str(rocScore))
def showPredictResults(model):
yPred = model.predict(xTest)
predProb = model.predict_proba(xTest)
print("### "+str(model)+" showMetrics ###")
showMetrics(yTest, yPred)
yPred = np.concatenate([predProb, yPred.reshape(-1, 1)], axis=1)
probPositivePred = yPred[:, 1]
print("### "+str(model)+" showPrecisionRecallCurve ###")
showPrecisionRecallCurve(yTest, probPositivePred)
print("### "+str(model)+" showRocCurve ###")
showRocCurve(yTest, probPositivePred)
print('##################################')
print('###### LogisticRegression ########')
print('##################################')
showPredictResults(gsLr)
print(' \n')
print(' \n')
print('##################################')
print('###### SVC ########')
print('##################################')
showPredictResults(gsSvc)
print(' \n')
print(' \n')
print('##################################')
print('######### RANDOM FOREST ##########')
print('##################################')
showPredictResults(gsRf)
print(' \n')
print(' \n')
print('##################################')
print('######### DecisionTree ###########')
print('##################################')
showPredictResults(gsDt)결과표 해석하는법 입니다.
-- 예제 --
### show_metrics ###
[[79 10]
[24 33]]
Acc : 0.7671232876712328
precision : 0.7674418604651163
recall : 0.5789473684210527
# 위의 결과에서
0 1
0 79 10
1 23 33
79 + 10 + 23 + 33 = 145 (test data 총 갯수 )
79: TN(true negative) / 실제 값이 0(negative)인데 0이라고 맞춘 갯수
10 : FN(false negative) / 실제 값이 0(negative)인데 1이라고 틀린 갯수
23: FP(false positive) / 실제 값이 1(positive)인데 0이라고 틀린 갯수
33 : TP(true positive) / 실제 값이 1(positive)인데 1이라고 맞춘 갯수
Acc는 전체 145개에서 79+33 맞춘 퍼센테이지이고
precision은 TP/(FP+TP)이며 33/(23+33) 입니다.
헬스케어 데이터에서는 질병이 걸렸느냐 안걸렸느냐, 이번 데이터에서는 30일 이내에 재방문 했느냐 안했느냐가 중요합니다.
따라서 1의 값을 얼마나 잘 맞추었냐가 더 중요합니다. 1의 값을 맞춘 비율이 precision입니다.
Acc로 판단했을때의 위험요소는 만약 1000개의 데이터에서 900개가 0이고 100개가 1일때(inbalance data)
1을 맞추지 않고 전부 0이라고 예측해버리면 90퍼센트의 정확도가 나오기 때문에 Acc로 판단하기는 위험합니다.
recall은 TP/(FN+TP)이며 1이라고 예측한 값들 중에 10과 33이 있으며 이중에서 얼마나 질병이 걸렸는지
잘 예측했는지 보는 결과입니다. 33/(10+33)
#- showPrecisionRecallCurve -
th val : [0.04994261 0.06008972 0.0710064 0.07128134]
precision(정확도) val : [0.44186047 0.4375 0.44094488 0.43650794]
recalls(재현율) val : [1. 0.98245614 0.98245614 0.96491228]
precisions, recall : 결론적으로 acc값보다 precision과 recall값이 더 의미가 있으며 모델 평가에 있어서 중요합니다.
# - showRocCurve -
* fpr : false positive rate
* tpr : true positive rate
* tpr = recall
thresholds :
thresholds를 만약 0.3으로 정했다면 예측값은 확률로 나오는데 만약 질병이 걸렸다고 예측하는 확률이
0.4라면 질병이 걸렸다고 1이라고 값을 반환합니다. 즉 thresholds는 걸렸다 안걸렸다를 확실하게 구분해주는
기준치라고 보면됩니다.
그럼 thresholds가 1이라면 당연히 0.99의 확률로 병에 걸렸다고 예측하여도 0의 값이 나옵니다.
show_precision_recall_curve의 그래프 교차지점의 thresholds의 값이 가장 적합한 기준치라고 봐도됩니다.
roc 커브는 이런 다양한 모델 평가 부분들을 한눈에 볼 수 있는 그래프 입니다. 30일 이내에 재방문하는 사람을
30일 이내에 재방문 할것이라고 예측하고 그렇지 않은 사람을 재방문 하지 않을 것이라고 예측하는 것이
tpr = 1 이고 fpr = 0이 되는 것입니다.
결국 roc 커브에서 모델의 평가가 좋다는 것은 커브의 밑면적 즉 auc의 넓이가 넓을수록 성능이 좋습니다.
roc_score값이 결국 예측을 얼마나 잘 했느냐입니다.
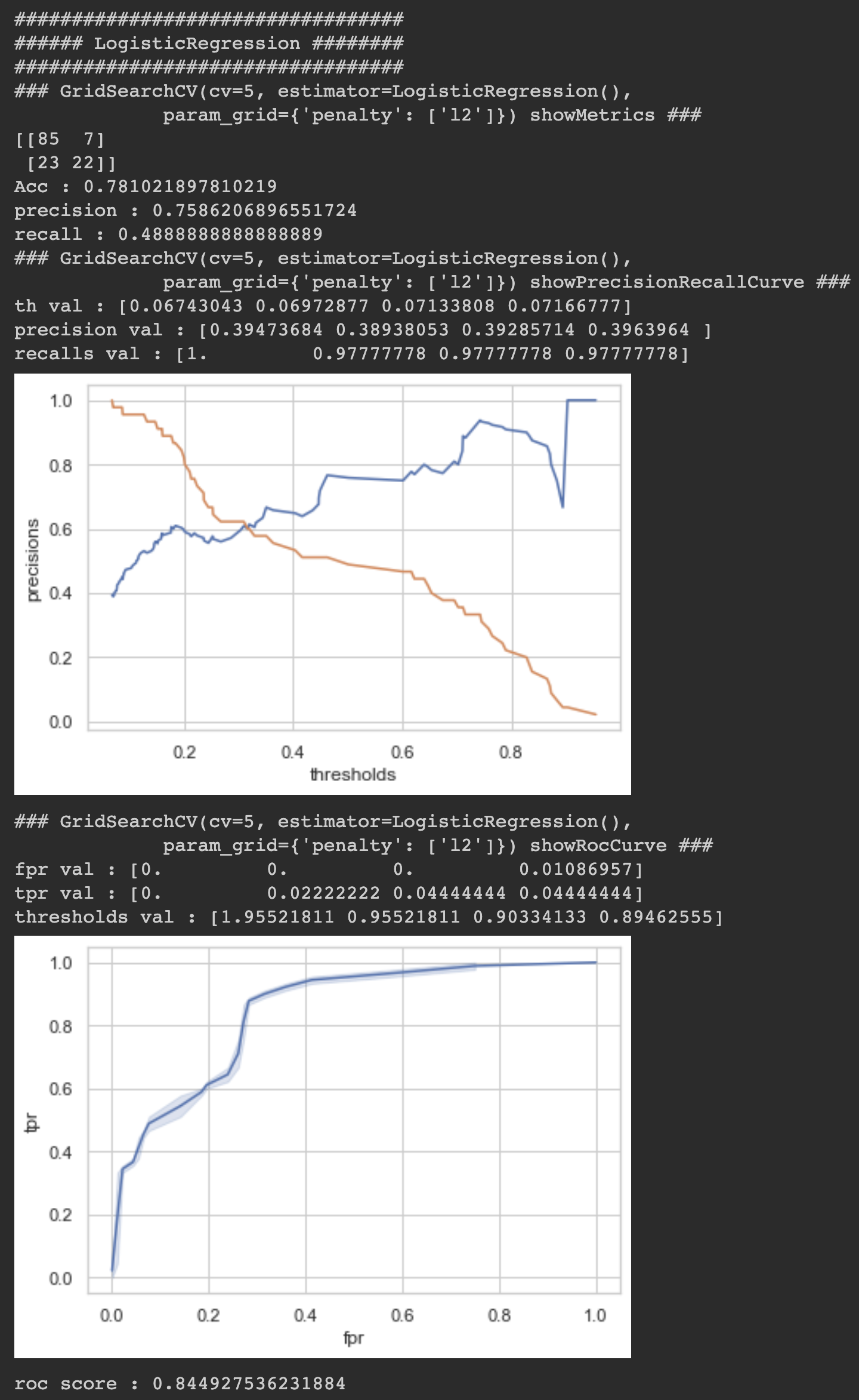
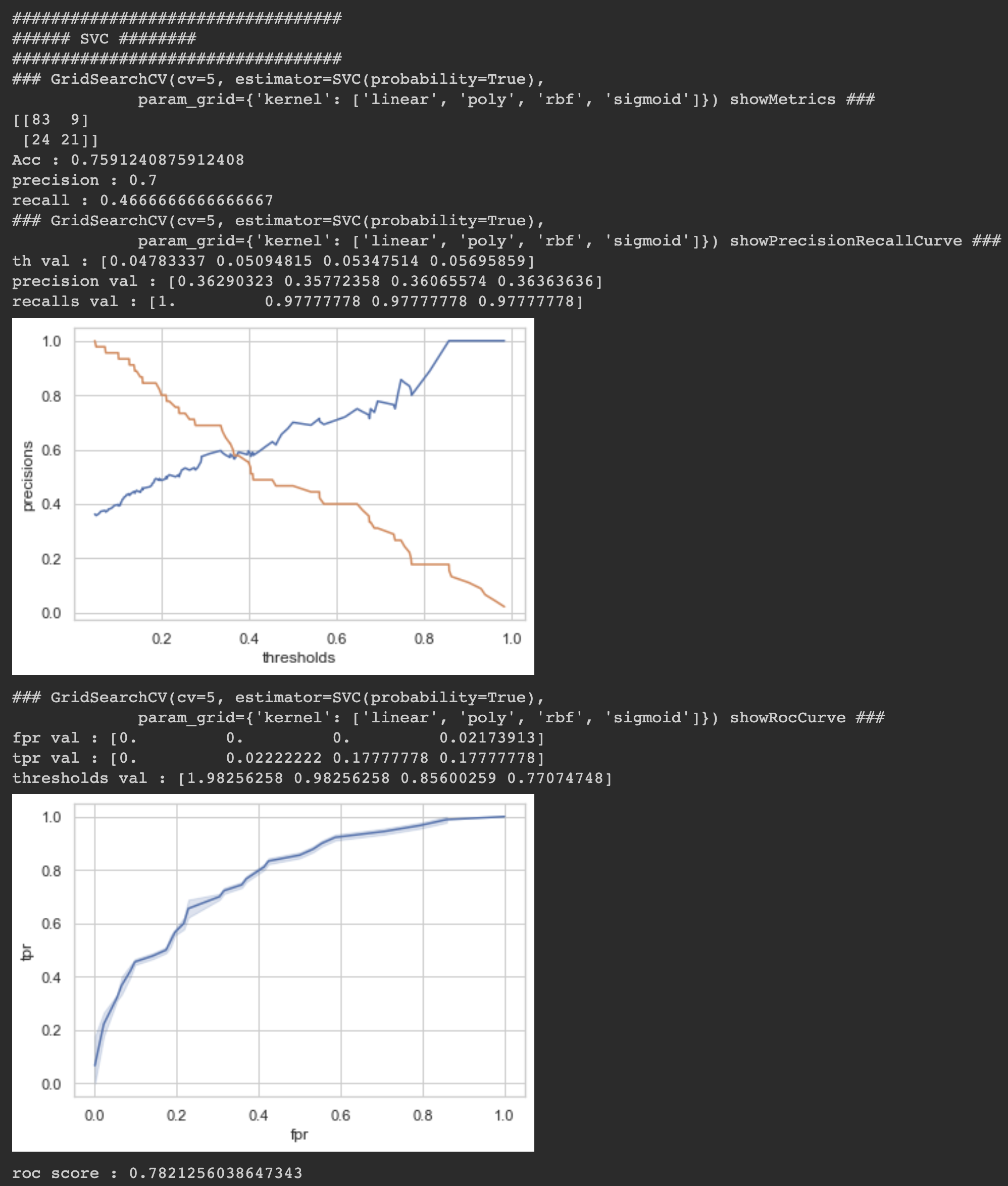
LogisticRegression ROC_SCORE : 84.4%
SVC ROC_SCORE : 78.2%
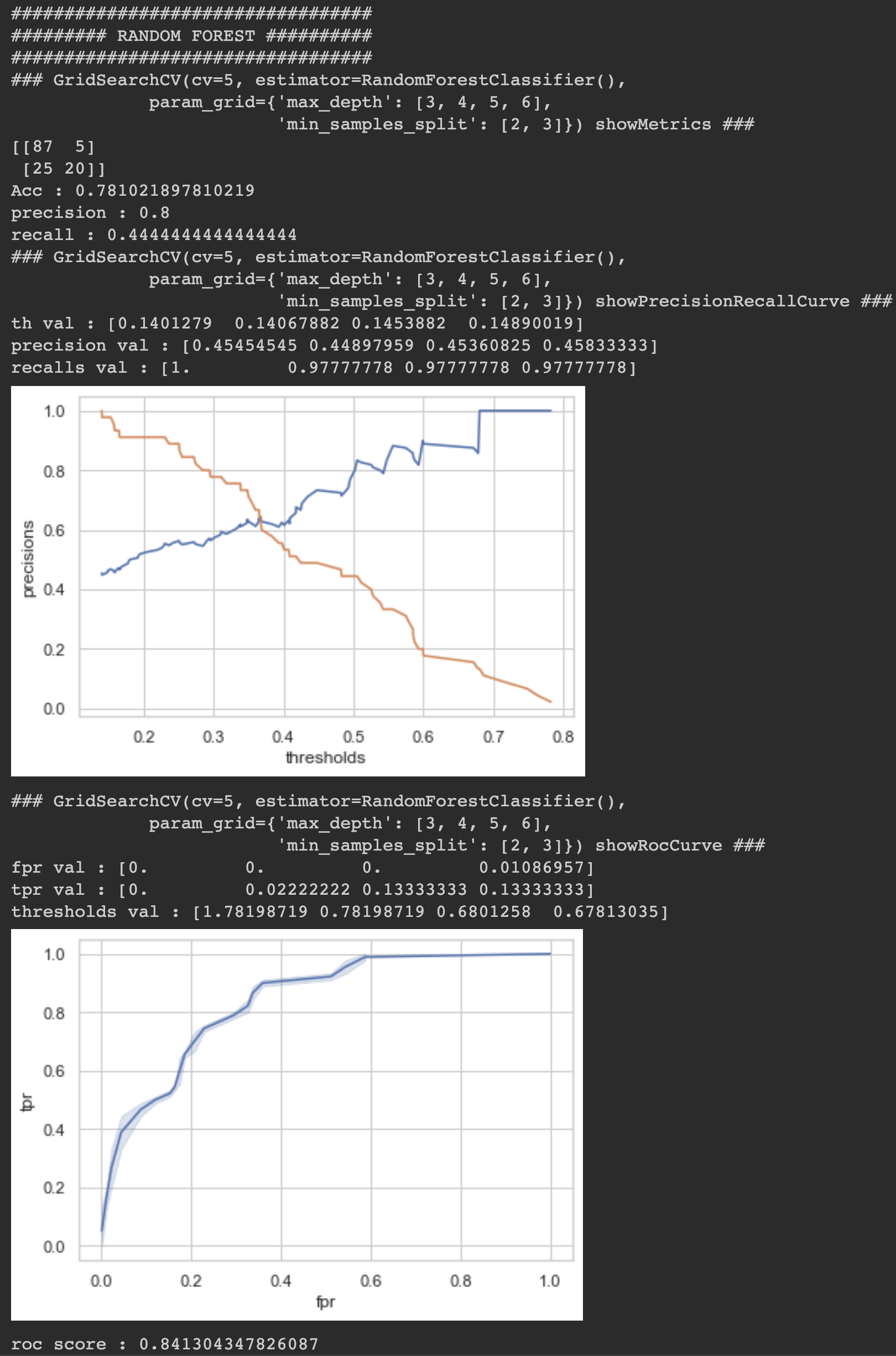
RandomForestClassifier ROC_SCORE: 84.1%
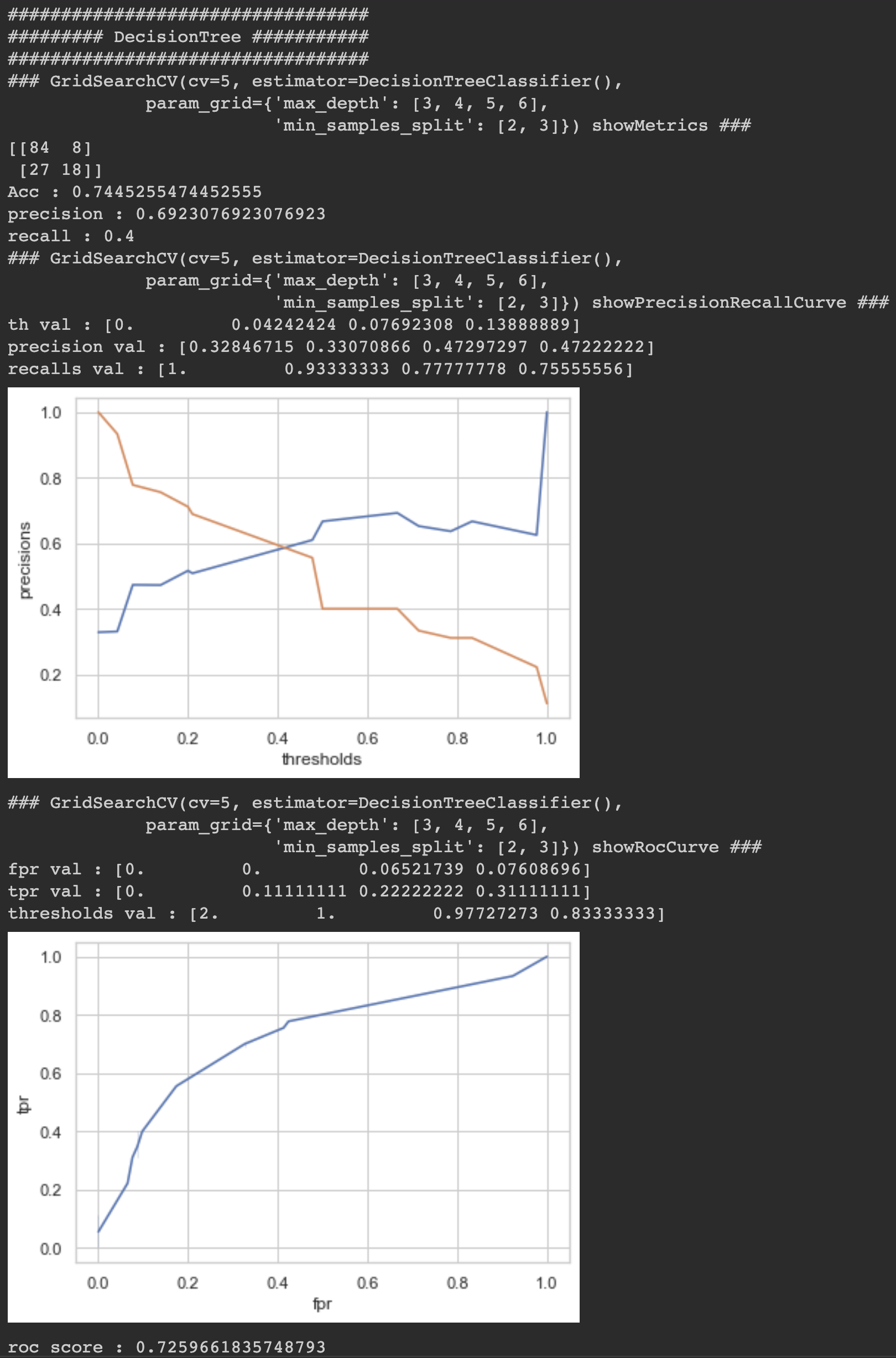
DecisionTreeClassifier ROC_SCORE : 72.5%
LogisticRegression이 84.4%로 가장 높게 나왔습니다.
자세한 소스코드는 아래의 깃헙 주소에서도 확인 가능합니다.
https://github.com/imtelloper/data-analysis-pimaindian-cls-clf.git
'AI > 인공지능 당뇨병 관련 예측(diabetes 130)' 카테고리의 다른 글
당신이 좋아할만한 콘텐츠
소중한 공감 감사합니다
포스팅 주소를 복사했습니다
이 글이 도움이 되었다면 공감 부탁드립니다.