Pima Indian Diabetes 데이터 Clustering & Classification (Part.1)
- -
- PROCESS -
데이터 탐색 -> 데이터 전처리 -> 데이터 클러스터링 -> 모델링 -> 성능 평가
이번에는 데이터분석의 입문용으로 좋은 pima indian diabetes 데이터로 군집화와 분류를 해보겠습니다.
코드는 git에 올려놨으니 참고하시면 되겠습니다.
https://github.com/imtelloper/data-analysis-pimaindian-cls-clf.git
데이터 출처 : https://www.kaggle.com/uciml/pima-indians-diabetes-database
############ 데이터 컬럼 설명 ############
Pregnancies: 임신 횟수
Glucose: 포도당 부하 검사 수치
BloodPressure: 혈압(mm Hg)
SkinThickness: 팔 삼두근 뒤쪽의 피하지방 측정값(mm)
Insulin: 혈청 인슐린(mu U/ml)
BMI: 체질량지수(체중(kg)/키(m))^2
DiabetesPedigreeFunction: 당뇨 내력 가중치 값
Age: 나이
Outcome: 클래스 결정 값(0 또는 1)
############ DOMAIN KNOWLEDGE ############
- 당뇨병 요인 -
약물 (당류 코르티코이드 등)을 투약 중인 경우
평소에 운동을 하지 않는 사람
이전에 공복혈당장애나 내당능장애를 진단 받았거나 당화혈색소가 5.7% 이상이었던 사람
중성지방이 높거나(> 250 mg/dL) 고밀도콜레스테롤이 낮은(< 35 mg/dL) 사람
심혈관 질환(뇌졸중, 관상동맥질환, 말초혈관질환)을 경험한 사람
※ 부모, 형제 중에 당뇨병을 가진 사람이 있는 경우
※ 임신성 당뇨병이 있었거나 4kg 이상의 아기를 분만한 여성
※ 혈압이 140/90 mmHg 이상이거나 항 고혈압 약물을 복용 중인 사람
※ 인슐린 저항성(고도 비만, 다낭성난소증후군, 흑색극세포증)이 있는 사람
############ 문제 파악 ############
outcome이 우리의 y 값이고 나머지는 x값이며 x값들을 받았을때 이 사람이 당뇨병에 걸렸는지 안걸렸는지
예측하는 문제이므로 분류 문제입니다.
# 데이터 탐색
우선 3개의 데이터를 뽑아봅니다. 이번 데이터는 전부 수치형이며 전처리할게 많이 없는 깔끔한 데이터 입니다.
데이터 분석의 입문용으로 시작하기 좋습니다.
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
import intelModule as imd
# intelModule은 자주쓰는 함수를 미리 만들어 놓은 클래스입니다.
im = imd.intelModule
df = pd.read_csv('./data/diabetes.csv')
df.info()
df.head(3)
자동 완성 기능을 편하게 쓰기위해 미리 변수에 데이터 컬럼명을 넣습니다.
pregnancies = "Pregnancies"
glucose = "Glucose"
bloodPressure = "BloodPressure"
skinThickness = "SkinThickness"
insulin = "Insulin"
bmi = "BMI"
diabetesPedigreeFunction = "DiabetesPedigreeFunction"
age = "Age"
outcome = "Outcome"데이터를 살펴 봅니다. 전체 768개이며 평균값, 표준 편차, 최소값, 최대값을 살펴봅니다.
df.describe()
# 참조 : https://seaborn.pydata.org/generated/seaborn.pairplot.html#seaborn.pairplot
# 혹시나 seaborn 버전이 안맞아서 에러가 난다면 참고하십시오.
sns.__version__
# seaborn 표 뒷배경을 화이트로 해야 글자가 잘 보입니다.
sns.set(style='whitegrid')
sns.pairplot(df)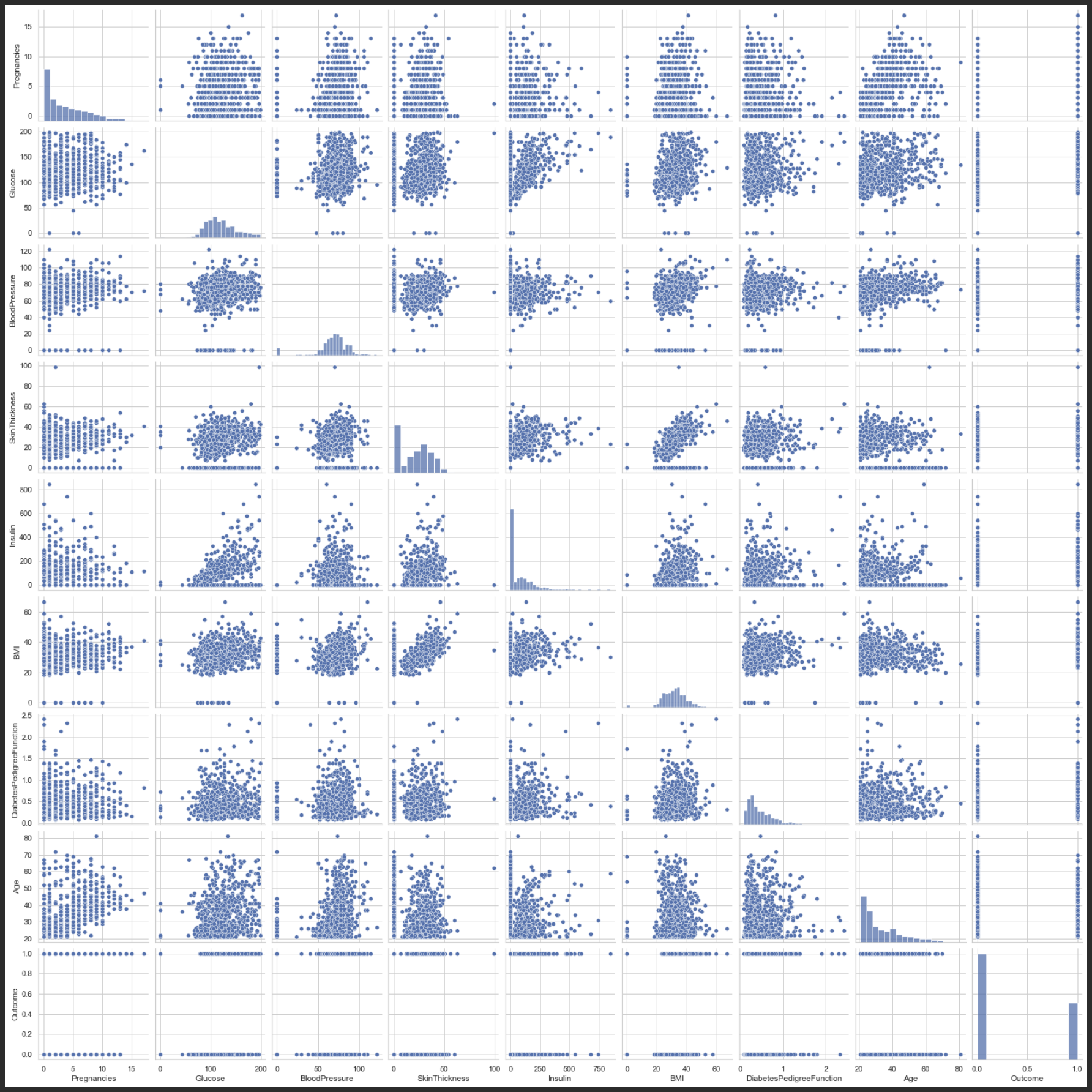
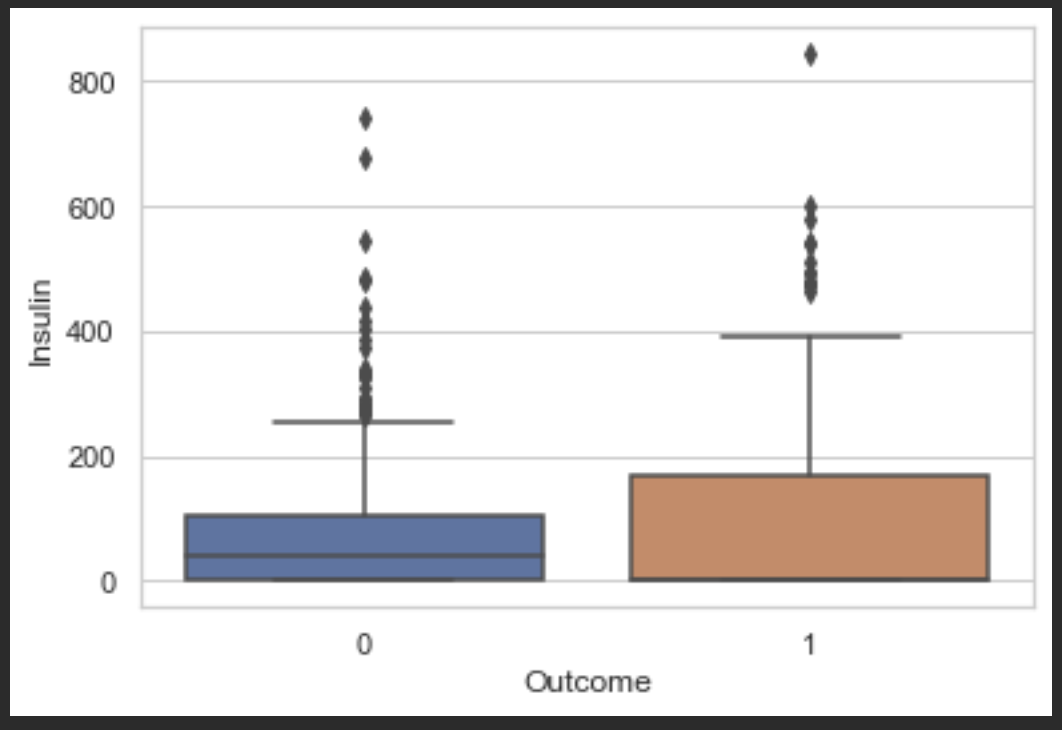
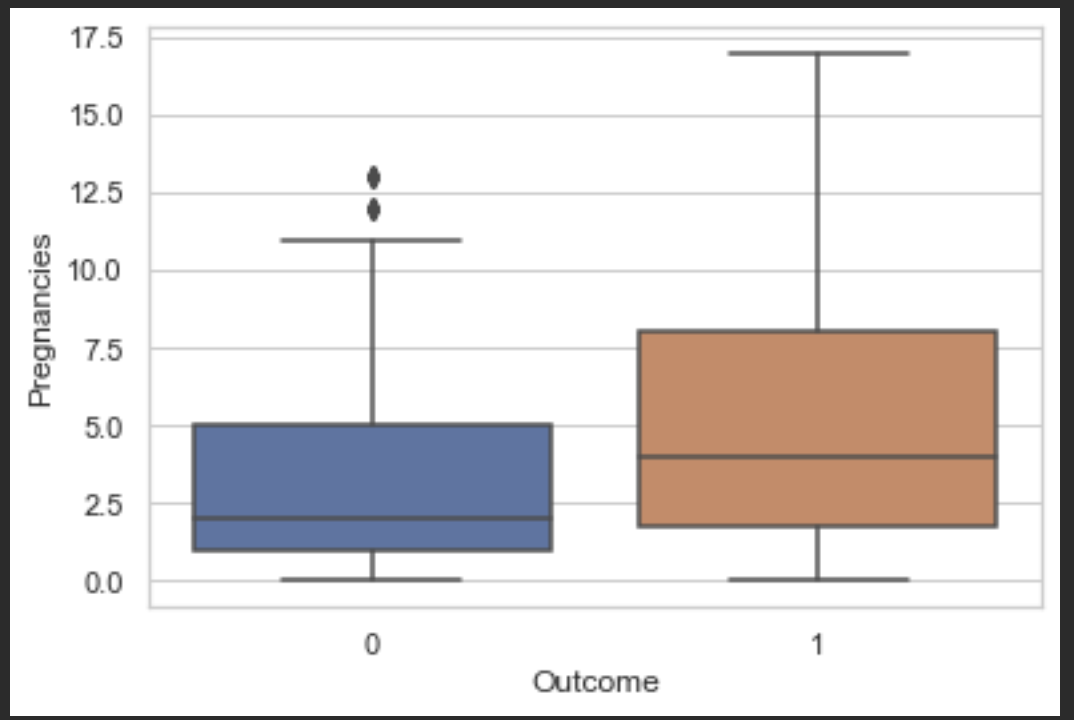
우선 전체적으로 박스플롯을 찍어보고 어떤 변수들이 의미 있는지 제거해야할 아웃라이어들이 있는 변수들을 파악해봅시다.
im.showBoxPlots(df, outcome)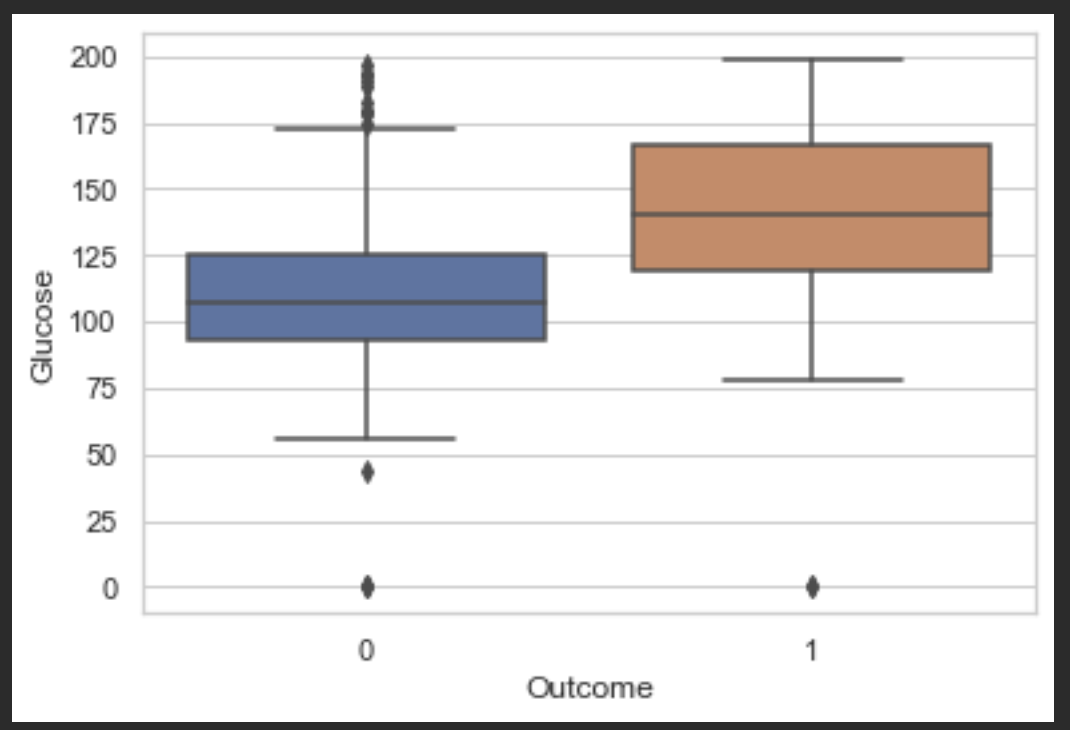
히트맵을 찍어봅니다.
Glucose가 0.47로 가장 당뇨병에 영향을 끼친다고 볼 수 있겠습니다.
plt.figure(figsize=(10, 8))
sns.heatmap(df.corr(), cmap='Wistia', annot=True)
plt.title('Heatmap for the Data', fontsize=20)
plt.show()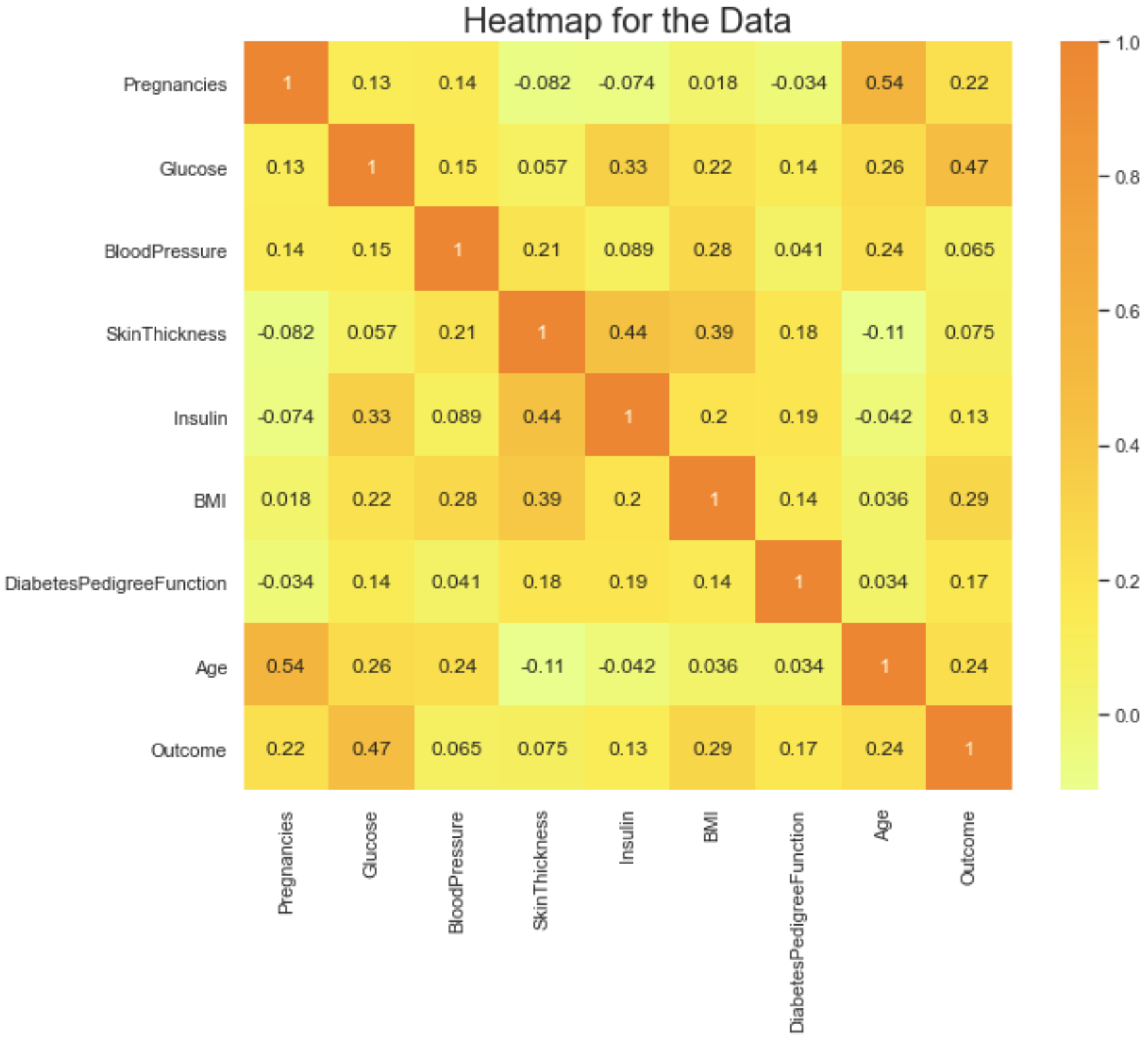
# 데이터 전처리
결측치가 있는지 한번 확인해 봅니다.
변수 모두 결측치가 없습니다.
df.isnull().sum()
임계값(Threshold)
애매한 값을 이분법으로 확실히 분류를 할 기준이 바로 임계값(Threshold)라고 합니다.
로지스틱 회귀 값을 이진 카테고리에 매핑(Mapping)하려면 분류 임계값(Classification Threshold, 결정 임계값이라고도 함)을 정의해야 합니다.
임계값보다 높은 값은 'true'를 나타내고 임계값보다 낮은 값은 'false'로 나타냅니다.
분류 임계값은 항상 0.5여야 한다고 생각하기 쉽지만 임계값은 문제에 따라 달라지므로 값을 조정해야 합니다.
이제 아웃라이어를 제거합니다. 아웃라이어란 데이터 상의 다른 값들의 분포와 비교했을때 비정상적으로 떨어져있는 관측치입니다.
예를들어 마라톤 대회 데이터가 있다고 하면 보통 사람들이 2시간에서 2시간 30분 사이에 다 완주를 하는데 대회에 그냥 참가
목적으로 온 사람들은 4시간, 5시간 뒤에 완주를 한 경우의 데이터 입니다.
일반적인 데이터가 아니기 때문에 학습에 도움이 안되므로 제거해 줍니다.
# 아웃라이어란 데이터 상의 다른 값들의 분포와 비교했을때 비정상적으로 떨어져있는 관측치입니다.
cols = df.columns
print("before drop outlier : {}".format(df.shape))
for col in cols:
# mean : 평균
mean = df[col].mean()
# std : 표준편차
std = df[col].std()
highThreshold = mean + 3 * std
lowThreshold = mean - 3 * std
# df[col] > threshold => true, false를 반환함 true면 1 false면 0
# 따라서 true인값만 sum을 하면 아웃라이어 값이 나옴
highNOutlier = np.sum(df[col] > highThreshold)
lowNOutlier = np.sum(df[col] < lowThreshold)
df.drop(df[df[col] > highThreshold].index[:], inplace=True)
df.drop(df[df[col] < lowThreshold].index[:], inplace=True)
df.dropna()
print("after drop outlier : {}".format(df.shape))
before drop outlier : (768, 9)
after drop outlier : (685, 9)
이번 데이터는 매우 깔끔한 데이터로 아웃라이어정도만 제거하고 전처리 해주겠습니다.
# 클러스터링
# 0번째 Pregnancies
# 1번째 Glucose
# 2번째 BloodPressure
# 3번째 SkinThickness
# 4번째 Insulin
# 5번째 BMI
# 6번째 DiabetesPedigreeFunction
# 7번째 Age
# 8번째 -1번째 Outcome
# clustering x : clx
clx = df.iloc[:, [1, -1]].values
print('clx', clx)
print(clx[0])
print(clx[1])
print(clx[2])
위의 HeatMap에서 봤듯이 Glucose가 가장 당뇨병과 상관 관계가 높아 Glucose와 당뇨병의 관계를 군집화 해보겠습니다.
from sklearn.cluster import KMeans
wcss = []
for i in range(1, 20):
kmeans = KMeans(n_clusters=i, init='k-means++')
kmeans.fit(clx)
wcss.append(kmeans.inertia_)
plt.plot(range(1, 20), wcss)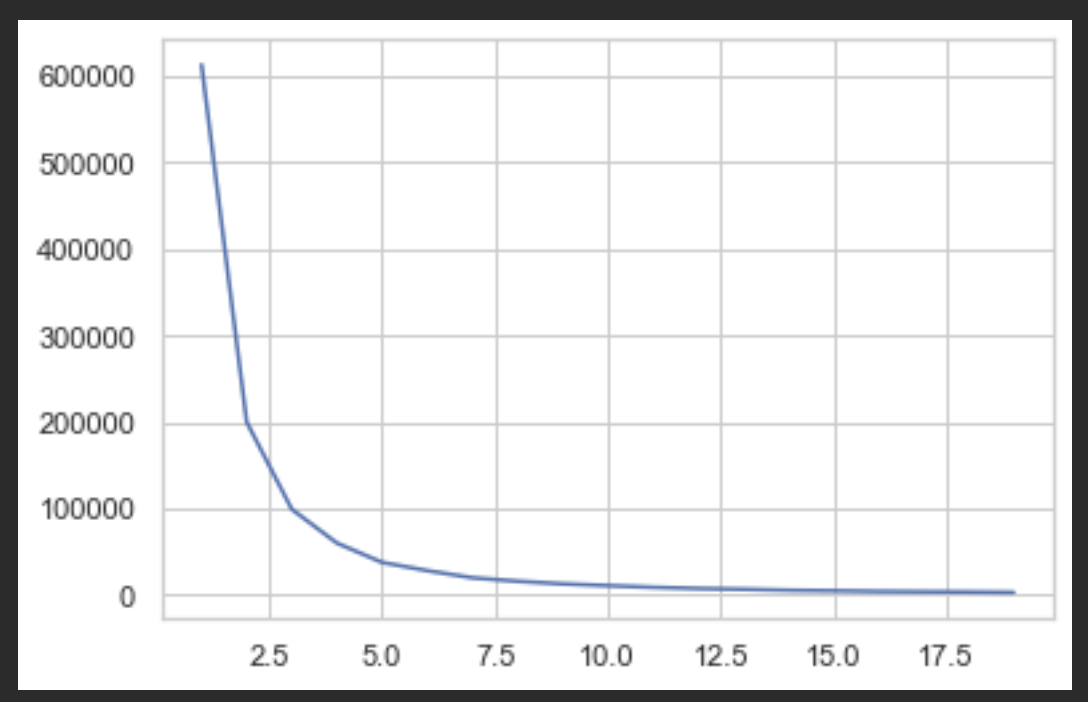
엘보우(팔꿈치처럼 접히는 부분이 최적의 n_clusters 갯수입니다.)
표를 보니 2.5에서 오른쪽으로 살짝 벗어난 3정도 되는곳이 적당해보입니다.
kmeans = KMeans(n_clusters=3, init='k-means++')
yPred = kmeans.fit_predict(clx)
print('yPred : ', yPred)K- 평균 클러스터링은 가장 간단하고 인기있는 비지도 머신 러닝 알고리즘 중 하나입니다.
일반적으로 비지도 알고리즘은 알려진 또는 레이블이 지정된 결과를 참조하지 않고 입력 벡터만 사용하여 데이터 세트에서 추론합니다.
K- 평균의 목표는 유사한 데이터 포인트를 함께 그룹화하고 기본 패턴을 발견합니다.
이 목표를 달성하기 위해 K- 평균은 데이터 세트에서 고정 된 수 (k)의 클러스터를 찾습니다.
클러스터는 특정 유사성 때문에 함께 집계 된 데이터 포인트 모음을 나타냅니다.
데이터 세트에 필요한 중심 수를 나타내는 목표 숫자 k를 정의합니다. 중심은 클러스터의 중심을 나타내는 가상 또는 실제 위치입니다.
클러스터 내 제곱합을 줄임으로써 모든 데이터 포인트가 각 클러스터에 할당됩니다.
즉, K- 평균 알고리즘은 k 개의 중심을 식별 한 다음 모든 데이터 포인트를 가장 가까운 클러스터에 할당하는 동시에 중심을 가능한 한 작게 유지합니다.
K- 평균의 '평균'은 데이터의 평균을 나타냅니다. 즉, 중심을 찾는 것입니다.
# 0번째 컬럼 즉 glucose 값들 중에서 클러스터링 분류 값이 0인 분류
print('Cluster-1 clx[yPred == 0, 0]', clx[yPred == 0, 0])
glucose0 = clx[yPred == 0, 0]
len(glucose0)
# 0번째 컬럼 즉 glucose 값들 중에서 클러스터링 분류 값이 1인 분류
print('Cluster-2 clx[yPred == 1, 0]', clx[yPred == 1, 0])
glucose1 = clx[yPred == 1, 0]
len(glucose1)
# 0번째 컬럼 즉 glucose 값들 중에서 클러스터링 분류 값이 2인 분류
print('Cluster-3 clx[yPred == 2, 0]', clx[yPred == 2, 0])
glucose2 = clx[yPred == 2, 0]
len(glucose2)
# 1번째 컬럼 즉 outcome 값들 중에서 클러스터링 분류 값이 0인 분류
print('clx[yPred == 0, 1]', clx[yPred == 0, 1])
outcome0 = clx[yPred == 0, 1]
len(outcome0)
# 1번째 컬럼 즉 outcome 값들 중에서 클러스터링 분류 값이 1인 분류
print('clx[yPred == 1, 1]', clx[yPred == 1, 1])
outcome1 = clx[yPred == 1, 1]
len(outcome1)
# 1번째 컬럼 즉 outcome 값들 중에서 클러스터링 분류 값이 2인 분류
print('clx[yPred == 2, 1]', clx[yPred == 2, 1])
outcome2 = clx[yPred == 2, 1]
len(outcome2)
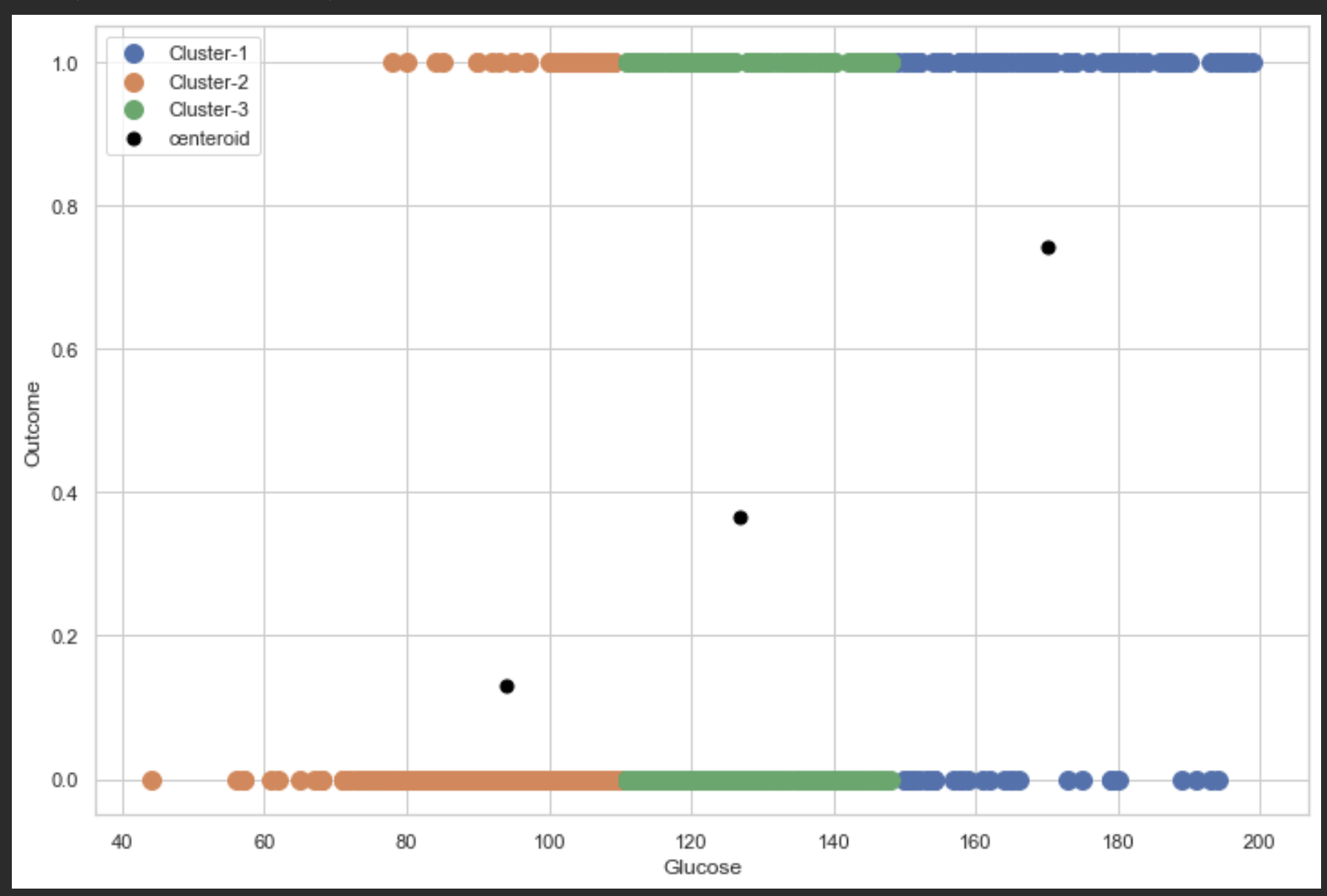
plt.figure(figsize=(12, 8))
plt.scatter(glucose0, outcome0, label='Cluster-1', s=100)
plt.scatter(glucose1, outcome1, label='Cluster-2', s=100)
plt.scatter(glucose2, outcome2, label='Cluster-3', s=100)
plt.scatter(kmeans.cluster_centers_[:, 0],
kmeans.cluster_centers_[:, 1],
s=50,
c='black',
label='centeroid')
plt.legend()
plt.xlabel(glucose)
plt.ylabel(outcome)우선 표만 보았을땐 Glucose가 높을수록 Outcome이 1인 집단이 많아 보입니다.
glucose의 평균과 각 집단의 총 갯수를 한번 찍어봅니다.
print('Cluster-1 glucose avg is : ' + str(np.mean(np.array(glucose0))) + ", and length " + str(len(outcome0)))
print('Cluster-2 glucose avg is : ' + str(np.mean(np.array(glucose1))) + ", and length " + str(len(outcome1)))
print('Cluster-3 glucose avg is : ' + str(np.mean(np.array(glucose2))) + ", and length " + str(len(outcome2)))

cluster0Column = [glucose, outcome]
cluster0 = pd.concat(
[pd.DataFrame(glucose0, columns=[glucose]), pd.DataFrame(outcome0, columns=[outcome])], axis=1)
print(cluster0)
cluster1Column = [glucose, outcome]
cluster1 = pd.concat(
[pd.DataFrame(glucose1, columns=[glucose]), pd.DataFrame(outcome1, columns=[outcome])], axis=1)
print(cluster1)
cluster2Column = [glucose, outcome]
cluster2 = pd.concat(
[pd.DataFrame(glucose2, columns=[glucose]), pd.DataFrame(outcome2, columns=[outcome])], axis=1)
print(cluster2)각 집단에서 당뇨병 걸린 사람의 퍼센트를 보았을때
glucose가 높은 집단이 퍼센트가 높은것을 확인할 수 있습니다.
이렇게 확인 후 특정 의미있는 집단만 추출하여 학습을 하여도 되겠습니다.
c0idx0 = cluster0[cluster0[outcome] == 0].index
c0idx1 = cluster0[cluster0[outcome] == 1].index
c1idx0 = cluster1[cluster1[outcome] == 0].index
c1idx1 = cluster1[cluster1[outcome] == 1].index
c2idx0 = cluster2[cluster2[outcome] == 0].index
c2idx1 = cluster2[cluster2[outcome] == 1].index
print('len(c0idx0)', len(c0idx0))
print('cluster1 집단에서 당뇨병 걸린 사람의 수 ', len(c0idx1))
print('cluster0Column', len(cluster0))
print('cluster1 집단에서 당뇨병 걸린 사람의 퍼센트 ', len(c0idx1) / len(cluster0))
print('len(c1idx0)', len(c1idx0))
print('cluster2 집단에서 당뇨병 걸린 사람의 수 ', len(c1idx1))
print('cluster0Column', len(cluster1))
print('cluster2 집단에서 당뇨병 걸린 사람의 퍼센트 ', len(c1idx1) / len(cluster1))
print('len(c2idx0)', len(c2idx0))
print('cluster3 집단에서 당뇨병 걸린 사람의 수 ', len(c2idx1))
print('cluster0Column', len(cluster2))
print('cluster3 집단에서 당뇨병 걸린 사람의 퍼센트 ', len(c2idx1) / len(cluster2))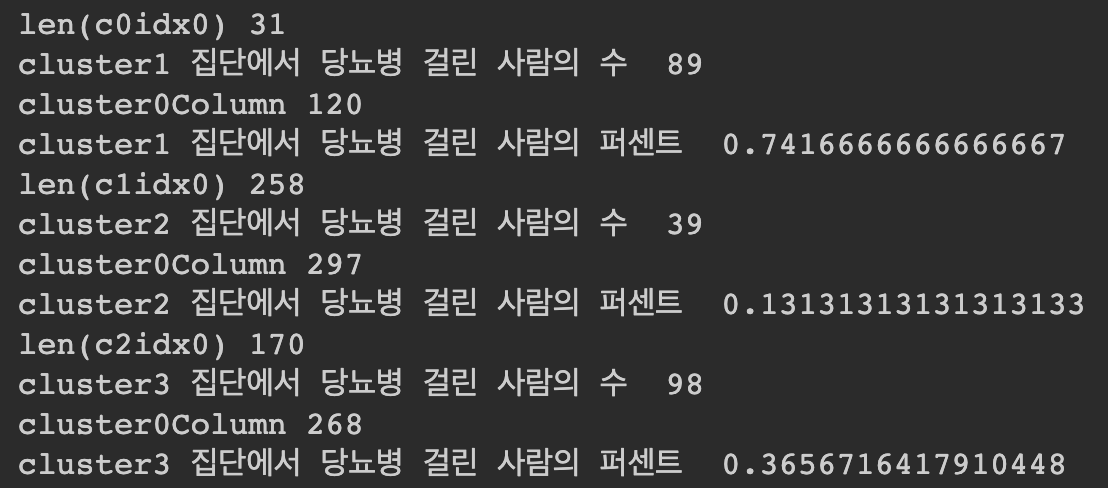
AgglomerativeClustering모델로 한번 더 클러스터링을 해주고 확인해봅니다.
from sklearn.cluster import AgglomerativeClustering
ac = AgglomerativeClustering(n_clusters=3, affinity='euclidean', linkage='ward')
yPred = ac.fit_predict(clx)
#visualizing clusters
plt.figure(figsize=(12, 8))
plt.scatter(clx[yPred == 0, 0], clx[yPred == 0, 1], label='Cluster-1', s=100)
plt.scatter(clx[yPred == 1, 0], clx[yPred == 1, 1], label='Cluster-2', s=100)
plt.scatter(clx[yPred == 2, 0], clx[yPred == 2, 1], label='Cluster-3', s=100)
plt.legend()
plt.xlabel(glucose)
plt.ylabel(outcome)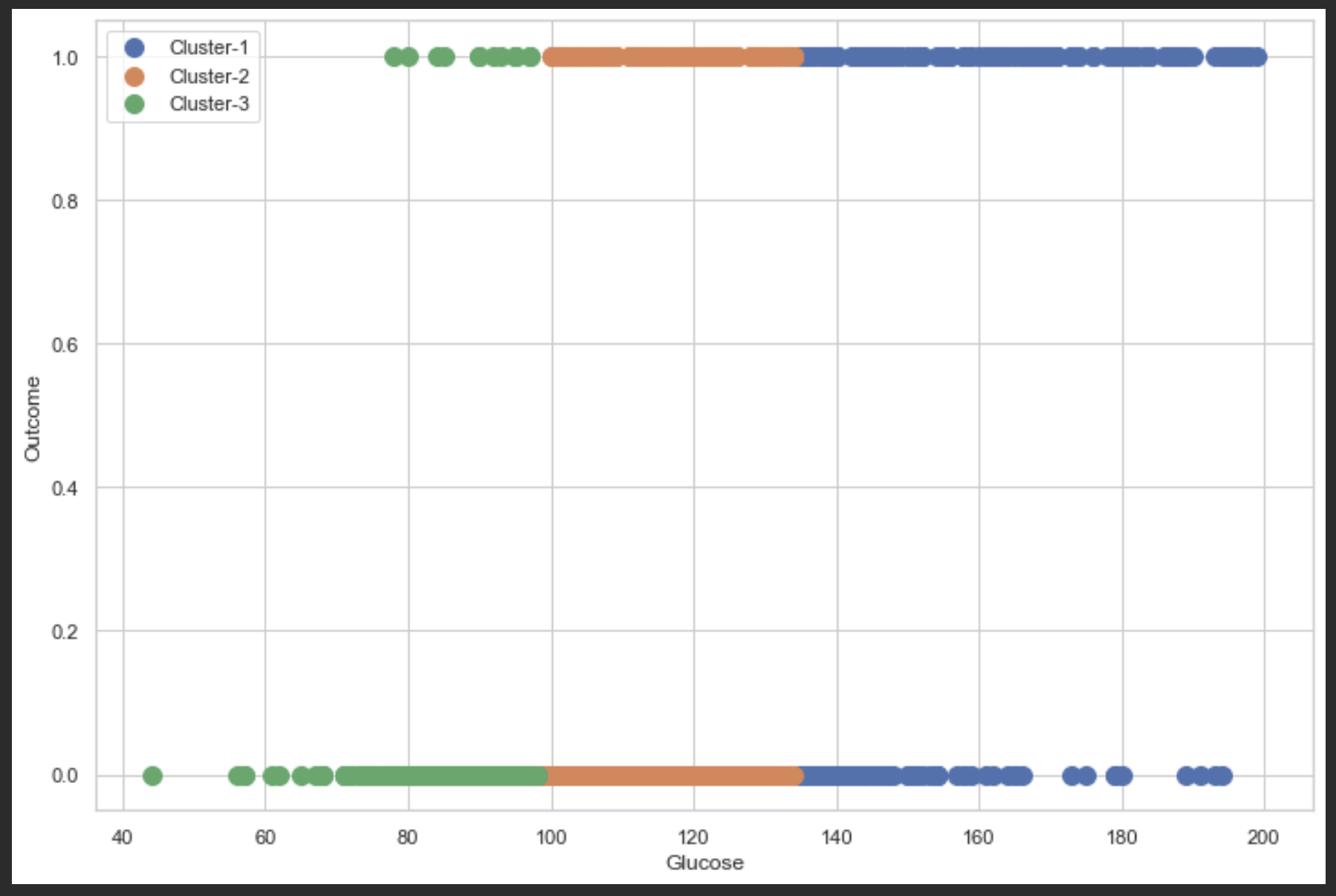
이만 클러스터링까지 마치고 다음으로 모델링과 성능평가를 해보겠습니다.
'AI > 인공지능 당뇨병 관련 예측(diabetes 130)' 카테고리의 다른 글
당신이 좋아할만한 콘텐츠
소중한 공감 감사합니다
포스팅 주소를 복사했습니다
이 글이 도움이 되었다면 공감 부탁드립니다.